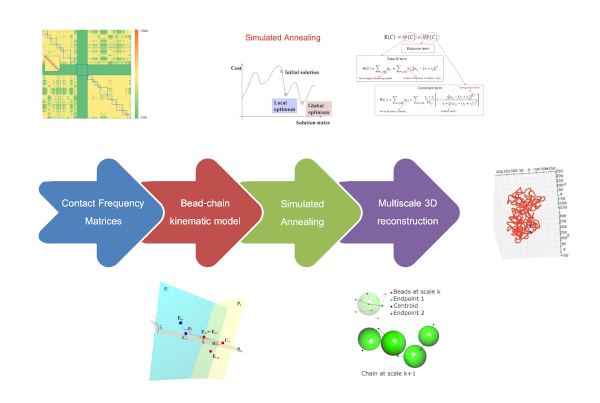
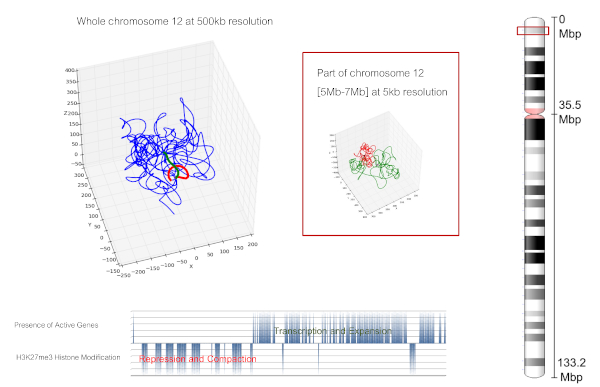
Il python-code ChromStruct fornisce una stima della struttura 3D della fibra di cromatina partendo da dati speraimentali di tipo Chromatin Conformation Capture (informazioni sui punti di contatto del DNA di popolazioni di milioni di cellule).
L'algoritmo adotta un approccio multiscala per esplorare la struttura dei cromosomi a diversi livelli di risoluzione e possiede una funzione di ottimizzazione di tipo bayesiano per esplorare lo spazio delle possibili configurazoni che la fibra di cromatina può assumere all'interno del nucleo compatibilmente con i constraints e i dati sperimentali. Il codice è open souce e dotato di una GUI attaraverso la quale è possibile settare tutti i parameri liberi.
La fibra è inizialmente divisa in segmenti indipendenti, le cui conformazioni vengono valutate separatamente ed in seguito modellate come singoli elementi di una fibra a più bassa risoluzione. L'algoritmo procede iterativamente fino al raggiungimento della minima risoluzione possibile. ChromStruct richiede in input solamente la matrice delle frequenze di contatto del DNA e restituisce come output le coordinate della catena a risoluzione piena, in formato txt e pdb.
Riferimenti:
Caudai C, Salerno E, Zoppe M, Tonazzini A. Estimation of the Spatial Chromatin Structure Based on a Multiresolution Bead-Chain Model. IEEE/ACM Transactions on Computational Biology and Bioinformatics. 2019;16(2):550-559.
Caudai C, Salerno E, Zoppe M, Merelli I, Tonazzini A. ChromStruct 4: A Python Code to Estimate the Chromatin Structure from Hi-C Data. IEEE/ACM Transactions on Computational Biology and Bioinformatics. 2019; 16: 1867-1878.
Caudai C, Salerno E, Zoppè M, Tonazzini A. Inferring 3D chromatin structure using a multiscale approach based on quaternions. BMC Bioinformatics. 2015;16:234.
Software:
E. Salerno, C. Caudai. CHROMSTRUCT v4.2 - Reconstruction of 3D chromatin structure from chromosome conformation capture data. Software, 2018, CNR-ISTI, Pisa, 2018-388694 DOI: 10.13140/RG.2.2.26123.39208.
Immagini: